Etablierung und Optimierung neuer Methoden
Für massenspektrometrie-basierte Proteomanalysen setzen wir hauptsächlich auf drei technische Bereiche, die die moderne, identifikationsorientierte und quantitative Proteomics repräsentieren: Shot-gun Proteomanalysen / datenabhängige Massenspektrometrie, SRM-basierte / zielgerichtete Massenspektrometrie und datenunabhängigen Massenspektrometrie. Für das Erreichen der maximalen Leistung sind wir stark vom technologischen Fortschritt der massenspektrometrischen Ausstattung abhängig. Um die diese moderne Ausstattung in angemessener Weise zu nutzen müssen Arbeitsabläufe optimiert oder neu entwickelt werden. Diese Bemühungen sind vor allem deshalb bedeutsam, weil das experimentelle Vorgehen häufig mehr über die wissenschaftliche Erkenntnisse entscheidet als die Geräte selbst.
- Becher et al. PMID: 23894095.
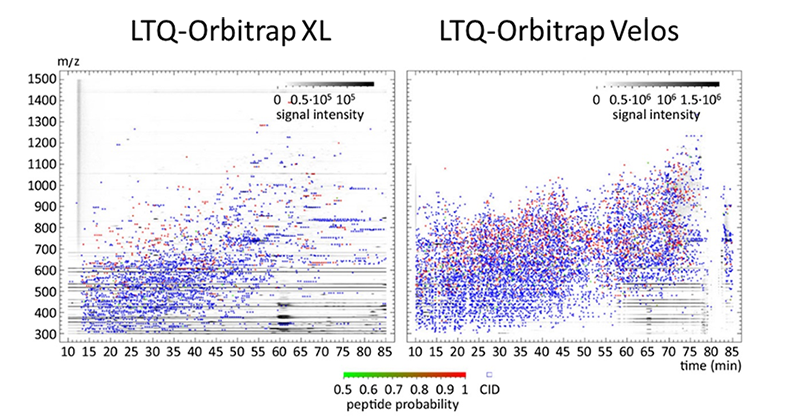
LC-MALDI
Trotz des Erfolges der ESI-basierten Massenspektrometrie in den letzten Jahren stellt die MALDI-MS für die Proteinidentifikation bei hohem Probendurchsatz eine zuverlässige und schnelle Technik in der klassischen Proteomanalyse dar. Eine weiterentwickelte Anwendung dient dem Nachweisen von Peptiden, die direkt von der LC-Säule eluiert auf eine MALDI-Platte aufgetragen und mittels MALDI-ToF/ToF identifiziert und quantifiziert werden. Auf diese Weise kann der LC-Lauf praktisch eingefroren werden. Diese Technik kann für spezielle analytische Aufgabenstellungen angewendet werden, wie z. B. für die Suche nach Peptiden die aufgrund ihrer speziellen Aminosäurezusammensetzung im Prozess der Elektronenspray-Ionisation benachteiligt werden.
- Hessling et al. PMID: 23788530.
- Hessling et al. PMID: 24161710.
Proteomweite Analyse posttranslationaler Modifikationen
Alle Organismen sind durch eine hohe Dynamik in der Proteinbiosynthese und ihrer Abbauprozesse charakterisiert. Durch zelluläre Herausforderungen (z.B. durch oxidativen Stress) verursachte posttranslationale Modifikationsereignisse, wie z.B. Phosphorylierungen, führen zu einer einzigartigen Regulation der Aktivität der in der Zelle vorhandenen Proteine. Wir arbeiten daraufhin, die molekularen Ziele dieser Modifikationen unter Verwendung von 2D-Gel basierten Top-down Proteomanalysen und durch umfangreiche Anreicherungstechniken für beispielsweise S/T/Y/R-Phosphorylierungen zu bestimmen.
- Elsholz et al. PMID: 22517742.
- Bäsell et al. PMID: 24457182.
Relative quantification approaches – 15N labeling, SILAC
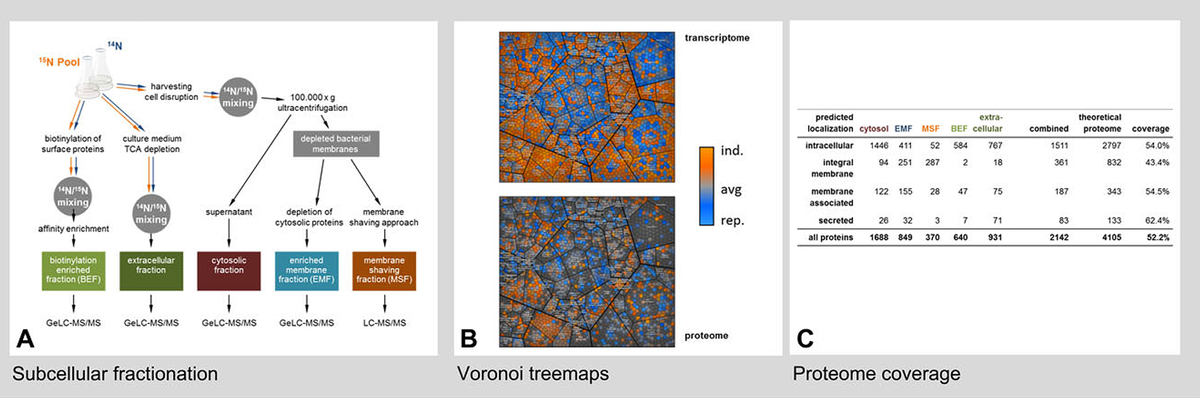
Wir haben einen leistungsfähigen Arbeitsablauf für die quantitative mikrobielle Proteomanalyse basierend auf der metabolischen Markierung mit einem schwerem Stickstoff-Isotop (15N) etabliert. Als Basis für die Bestimmung der relativen Mengen dient der Vergleich sequenzidentischer Peptide, die mit unterschiedlichen Isotopen aufgebaut sind. Diese Technik ist bei vor allem bei solchen Mikroorganismen leicht einsetzbar, die auf definierten (minimalen) Medien wachsen können. Komplexe Medien können dann verwendet werden, wenn diese durch den Austausch aller biologisch-aktiver Stickstoffquellen mit ihren schweren Isotopen modifizierbar sind. Wir nutzen beide beschriebene Ansätze um unterschiedliche Mikroorganismen zu kultivieren und damit 15N-markierte Proteomproben zu erzeugen. Das Minimalmedium mit schwerem Stickstoffsalz ((NH4)2SO4 oder (NH4)Cl) oder das komplexe Medium (basierend auf markierten Algen) kann für die relative Quantifizierung in der Proteomanalyse genutzt werden.
- Otto et al. PMID: 24376008.
- Dreisbach et al. PMID: 18491319.
- Becher et al. PMID: 19997597.
- Otto et al. PMID: 21266987.
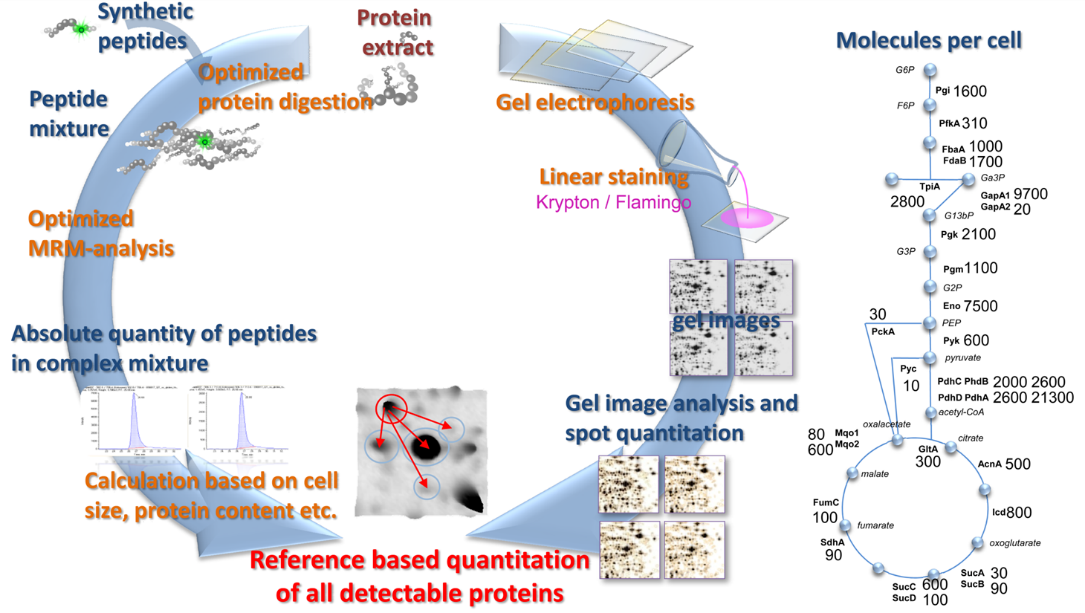
Absolute Proteinquantifizierung
Die absolute Quantifizierung, die vor allem in systembiologischen Studien verfolgt wird, stellt die anspruchsvollste technische und biologische Fragestellung in der Proteomics dar. Für die umfassendsten und genauesten Datensätze werden absolute Proteinmengen benötigt, die in Kombination mit anderen „Omics“-Daten einen tiefen modellhaften Einblick in die intrazellulär regulierten Prozesse ermöglichen.
In unseren Laboren werden beispielsweise diese Methoden verwendet:
Zielgerichtete Analyse von einzelnen Proteinen (selected reaction monitoring, SRM)
Für die absolute Quantifizierung ist es besonders wichtig die endogenen Peptide einer Proteomprobe mit isotopenmarkierten, zugemischten Peptiden einer bekannten Konzentration zu vergleichen. Wir verwenden den AQUA-Ansatz (absolute Quantifizierung) regelmäßig um einzelne Proteine, z. B. Mitglieder kleiner Proteinkomplexe, zu bestimmen oder global in Kombination mit 2D-Gel basierten Ansätzen das Proteom absolut zu quantifzieren.
- Delumeau et al. PMID: 21710567.
- Maass et al. PMID: 21395229.
- Muntel et al. PMID: 24696501.
SRM-2D PAGE
Für die absolute Quantifizierung haben wir zwei gut bekannte und weit verbreitete Proteomtechniken kombiniert: die Trennung der Proteine mittels 2D-Gel-Elektrophorese und die absolute Quantifizierung der Peptide durch AQUA auf einem Triple Quadrupol MS. Die Bestimmung der absoluten Mengen sogenannter „Ankerproteine“ auf 2D-Gelen ermöglicht uns die Kalibrierung aller anderen Proteine im gleichen Gel. Dies ist aktuell die kostengünstigste Methode um die absoluten Proteinmengen einiger hundert Proteine und ihrer Isoformen in einem komplexen Proteom zu bestimmen.
- Maass et al. PMID: 21395229.
- Otto et al. PMID: 24376008.
MSE DIA
Der Arbeitsablauf für die zweite absolute Quantifizierungsmethode basiert auf der MSE-Technologie, die sich für die fehlerfreie Identifikation und die Quantifizierung von komplexen Proteomproben eignet. Das Hauptmerkmal der Methode ist die datenunabhängige Akquirierung der Vorläuferionen für die MS/MS. Absolute Daten werden durch den Vergleich der drei Peptiden der höchsten Intensität eines jeden Proteins gegen einen Standard bekannter Konzentration abgeleitet (Hi3-Methode).
- Muntel et al. PMID: 24696501.
